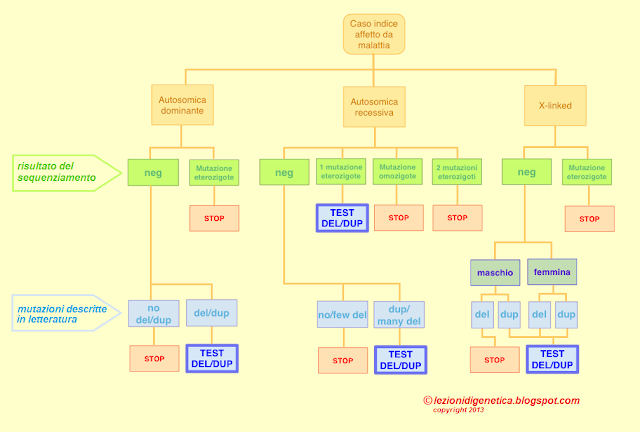
L'approccio di analisi genetica molecolare tipico prevede per la stragrande maggioranza dei geni il sequenziamento e il l'analisi di delezione/duplicazione (tramite MLPA o qPCR). L'analisi di delezione/duplicazione in realtà non è sempre indicata. Nella maggior parte dei casi la mutazione malattia consiste in una mutazione puntiforme (mutazione di un singolo nucleotide) o in un piccola delezione/inserzione/duplicazione di poche nucleotidi che possono alterare o meno il frame di lettura. Per questo genere di mutazione il sequenziamento diretto su filamento singolo e doppio (tramite metodica Sanger o NGS) rappresenta l'approccio migliore. Non raramente, tuttavia, la mutazione malattia consiste in una grande delezione o duplicazione di centinaia o migliaia di nucleotidi che tramite il sequenziamento non è rilevabile (o non direttamente, almeno). Prendiamo l'esempio di una malattia a trasmissione autosomica dominante, nella quale ci si aspetta di trovare una mutazione su un cromosoma soltanto. Tipicamente, nel caso di mutazione puntiforme, questa verrà rilevata in eterozigosi. Se però il paziente è affetto come conseguenza della presenza di una grossa delezione di parte o dell'intero gene su un cromosoma, il sequenziamento fallirà nel rilevarla. Il sequenziamento, infatti, analizza gli amplificati del gene che sono stati precedentemente ottenuti con la reazione di polimerizzazione a catena (polymerase chain reaction, PCR). Se una porzione di gene è assente su un cromosoma, i primer della PCR si legheranno comunque all'altro cromosoma, producendo sempre un amplificato. Al sequenziamento (che non è una metodica in grado di distinguere realmente i due cromosomi) non otterremo quindi la sequenza del frammento deleto (vedremo in effetti solo quella del cromosoma sano). Se al sequenziamento non viene rilevata alcuna mutazione, dunque, in caso di malattia a trasmissione autosomica dominante può essere indicata l'analisi di delezione. In realtà questo genere di mutazioni non sempre è atteso in tutti i geni. Tipicamente le malattie a trasmissione autosomica dominante sono caratterizzate da mutazioni 'gain of function', mentre le delezioni sono mutazioni non-funzionali che aboliscono il trascritto (e quindi la proteina) piuttosto che dare origine a una proteina con funzionalità aumentata o modificata. Tuttavia anche alcune malattie a trasmissione autosomica dominante possono essere causate da alleli non funzionali e, in caso di sequenziamento negativo, il test di delezione (tramite MLPA o qPCR) può quindi essere indicato.
Ma veniamo ora alle malattie a trasmissione autosomica recessiva, nelle quali gli alleli non funzionali (mutazioni non-senso, frame-shifting e grandi delezioni) sono piuttosto frequenti. Nelle malattie a trasmissione autosomica recessiva ci aspettiamo due mutazioni: una su ciascun cromosoma. Le mutazioni possono quindi essere in omozigosi (la stessa identica mutazione è cioè presente su entrambi i cromosomi) o in eterozigosi composta (i due cromosomi recano ciascuno una mutazione diversa, ma ugualmente patogenica). L'omozigosi o l'eterozigosi composta per le mutazioni puntiformi o per piccole delezioni/inserzioni/duplicazioni è visibile al sequenziamento. L'omozigosi per una grande duplicazione è invece sospettabile dal fallimento della PCR prima del sequenziamento. In altri termini, mancando il gene (o parte del gene) su entrambi i cromosomi, i primer della PCR non trovano niente a cui legarsi e non producono quindi alcun amplificato. Risultato: il sequenziamento risulta impossibile. In caso di eterozigosi composta per una mutazione puntiforme e una grande delezione invece, ecco che la grande delezione non è rilevabile al solo sequenziamento perchè presente allo stato eterozigote. Per contro, sia in caso di eterozigosi che in caso di omozigosi le grandi duplicazioni non sono mai rilevabili al sequenziamento. Esiste poi il capitolo delle malattie a trasmissione X-linked, che richiede discorsi separati per maschi e femmine. Infatti, essendo il maschio portatore di un solo cromosoma X, una grande delezione emizigote è sospettabile nel maschio dalla mancata amplificazione (fallimento della PCR prima del sequenziamento). Non così nelle femmine, dove i cromosomi X sono due, e nei maschi affetti da sindrome di Klinefelter, che pure hanno due cromosomi X. Anche nel caso di ereditarietà X-linked, sia che si tratti di femmine o di maschi, le grandi duplicazioni non sono mai rilevabili dal sequenziamento.
Dunque, riassumendo:
1. Malattia a trasmissione autosomica dominante:
- sequenziamento positivo: analisi di delezione/duplicazione non indicata
- sequenziamento negativo e mutazioni non funzionali non attese: analisi di delezione/duplicazione non indicata
- sequenziamento negativo e mutazioni non funzionali attese (o già descritte in letteratura): analisi di delezione/duplicazione indicata
2. Malattia a trasmissione autosomica recessiva:
- sequenziamento negativo: analisi di delezione/duplicazione non indicata
- sequenziamento positivo per un'unica mutazione: analisi di delezione/duplicazione indicata
- sequenziamento positivo per una mutazione omozigote o per due mutazioni in eterozigosi composta: analisi di delezione/duplicazione non indicata
3. Malattia a trasmissione X-linked:
> in un maschio:
- sequenziamento positivo: analisi di delezione/duplicazione non indicata
- sequenziamento negativo e duplicazioni non descritte: analisi di delezione/duplicazione non indicata
- sequenziamento negativo e duplicazioni descritte: analisi di delezione/duplicazione indicata.
> in una femmina:
- sequenziamento positivo: analisi di delezione/duplicazione non indicata