Quando una variante è riportata nel database dei polimorfismi di singolo nucleotide dell'NCBI (dbSNP) si è tentati di pensare che questa sia sicuramente un polimrofismo e, in quanto tale, innocua dal punto di vista clinico. Ciò non corrisponde al vero, in quanto in dbSNP sono a volte riportate anche mutazioni-malattia vere e proprie. A volte si tratta di mutazioni confermate in letteratura (ciò può essere evidente nelle prime righe della entry), altre volte si tratta semplicemente di varianti con frequenze alleliche molto basse, non ancora caratterizzate funzionalmente, ma di fatto patogeniche. La frequenza allelica è in effetti la prima caratteristica da valutare. In alcuni casi è la frequenza allelica riportata è molto bassa, in altri casi è addirittura non riportata. È necessario anzitutto ricordare che la frequenza allelica può essere riportata in punti diversi della pagina di dbSNP. In condizioni ideali si troverà la MAF (Minor Allele Frequency) in aggiunta ai valori di frequenza allelica e genotipica riscontrati in gruppi di campioni diversi (si tratta delle entry ss) e ai valori di frequenza allelica riscontrati nelle diverse etnie dello studio 1000 Genomes. A volte la MAF manca, ma sono comunque presenti alcune ss e/o i valori di 1000 Genomes. In altri casi, manca tutto, fatto che può essere considerato indicativo di una frequenza allelica molto bassa nella popolazione generale.
Vediamo ad esempio la variante rs377722397 nel gene SCN5A (le cui mutazioni possono causare la
sindrome di Brugada o la cardiomiopatia dilatativa). Per questa variante mancano del tutto i valori di MAF, i valori sulla diversità di popolazione e anche i dati dello studio 1000 Genomes. Parte della schermata dbSNP di questo SNP è riportata in figura 1.
 |
| Figura 1: variante rs377722397 del gene SCN5A: manca del tutto l'informazione sulla frequenza allelica e genotipica (MAF source, population diversity). La schermata di 1000 Genomes non è rappresentata. |
Vediamo un altro esempio nello stesso gene: rs201832649, una transizione A>G predetta generare una variante missenso p.Asn1659Asp in NP_001092874.1 (figura 2). Per questa variante si dispone della MAF dedotta dallo studio 1000 Genomes. Come evidenziato in figura, la frequenza allelica di questa variante nelle diverse popolazioni è stimata essere molto bassa.
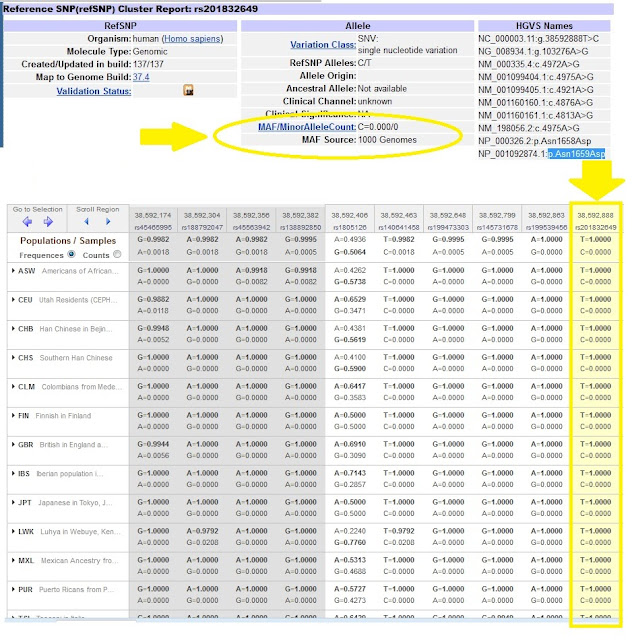 |
| Figura 2: esempio di variante allelica del gene SCN5A con frequenza allelica estremamente bassa (rs201832649). La parte inferiore della figura illustra la schermata relativa ai dettagli dello studio 1000 Genomes: si noti la frequenza molto bassa della variante rara (0.0000) nelle diverse etnie. |
Vediamo ora nello stesso gene un altro esempio, nel quale sono disponibli anche i dati sulla diversità di popolazione (population diversity), forniti dalle entry ss#: rs80197373, una trasversione localizzata profrondamente nell'introne (c.4814-1347A>T in NM_001099404.1). Questa variante ha una MAF di 0.068, ossia una frequenza allelica del 6,8%. La MAF è tuttavia solo un valore medio di quanto rilevato nell diverse popolazioni dello studio 1000 Genomes. E infatti, come è evidente osservando la figura 3, la frequenza allelica di questa variante cambia notevolemente da popolazione a popolazione. I dati della diversità di popolazione di questa variante (population diversity) non si discostano molto dai valori di 1000 Genomes.
 |
| Figura 3: variante intronica rs80197373 del gene SCN5A. La MAF (0.068) è
un valore medio di tutte le frequenze alleliche riscontrate nelle
diverse etnie dello studio 1000 Genomes. Infatti, questa variante
risulta avere una frequenza allelica inferiore negli americani di
origine africana (a), ma molto superiore nei cinesi (b, c). Nei britannici, invece, questa variante risulta essere assai rara (d). La sezione "Population Diversity" fornisce qualche infromazione in più. Ad esempio, nel campione di soggetti ss239208213, costituito da 120 cromosomi (quindi 60 persone), la frequenza allelica di A è 11,7%. |
Nota: per visualizzare i dettagli delle frequenze alleliche nelle varie popolazioni di 1000 Genomes asta cliccare sul simbolo qui sotto, che si trova nella schermata principale dello SNP:
Per un esempio, potete guardare anche
QUESTO VIDEO sul nostro canale Youtube.
.jpg)





Nessun commento:
Posta un commento